Monografia: Valutazione e comunicazione del rischio in Sicurezza alimentare
 E’ stato recentemente pubblicata dalla Milano University Press la monografia “Valutazione e Comunicazione del rischio“ a cura della Prof.ssa Maria Longeri. Il volume, scritto da specialisti ed esperti della materia, rappresenta una guida per coloro che si avvicinano per la prima volta al tema della valutazione del rischio in sicurezza alimentare. Il testo descrive le metodologie, il corpo normativo, le conoscenze e le competenze necessarie per effettuare un valutazione e comunicazione del rischio efficace nell’ambito della sicurezza alimentare.
E’ stato recentemente pubblicata dalla Milano University Press la monografia “Valutazione e Comunicazione del rischio“ a cura della Prof.ssa Maria Longeri. Il volume, scritto da specialisti ed esperti della materia, rappresenta una guida per coloro che si avvicinano per la prima volta al tema della valutazione del rischio in sicurezza alimentare. Il testo descrive le metodologie, il corpo normativo, le conoscenze e le competenze necessarie per effettuare un valutazione e comunicazione del rischio efficace nell’ambito della sicurezza alimentare.
Fonte: IZS Lazio Toscana
 Promuovere l’uso responsabile degli antimicrobici negli animali è uno degli obiettivi principali del Regolamento sui Prodotti Medicinali Veterinari che è diventato applicabile nell’Unione Europea il 28 gennaio 2022. Tra le misure introdotte dal Regolamento per combattere la resistenza agli antimicrobici ci sono restrizioni sull’uso profilattico dei farmaci antimicrobici negli animali. Completato il
Promuovere l’uso responsabile degli antimicrobici negli animali è uno degli obiettivi principali del Regolamento sui Prodotti Medicinali Veterinari che è diventato applicabile nell’Unione Europea il 28 gennaio 2022. Tra le misure introdotte dal Regolamento per combattere la resistenza agli antimicrobici ci sono restrizioni sull’uso profilattico dei farmaci antimicrobici negli animali. Completato il 
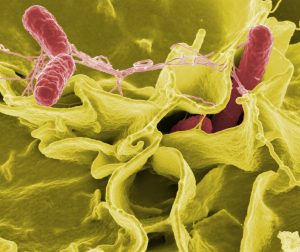 Tra gennaio e ottobre del 2023 14 Paesi dell’UE/SEE, il Regno Unito e gli Stati Uniti hanno segnalato 335 casi legati a questo focolaio epidemico.
Tra gennaio e ottobre del 2023 14 Paesi dell’UE/SEE, il Regno Unito e gli Stati Uniti hanno segnalato 335 casi legati a questo focolaio epidemico.

 Un pacchetto di 13 interventi per guidare la definizione delle priorità dei paesi nello sviluppo, nell’attuazione e nel monitoraggio dei piani d’azione nazionali sulla resistenza antimicrobica (AMR).
Un pacchetto di 13 interventi per guidare la definizione delle priorità dei paesi nello sviluppo, nell’attuazione e nel monitoraggio dei piani d’azione nazionali sulla resistenza antimicrobica (AMR). Finché si tratta di esseri umani o di animali da allevamento, la sfida può anche essere vinta. Ma quando l’epidemia spicca il volo, letteralmente, la circolazione del virus diventa ufficialmente fuori controllo. È quello che è accaduto con l’influenza aviaria causata da un sottotipo del virus H5N1 che dal 2020 sta facendo strage di volatili in tutto il mondo.
Finché si tratta di esseri umani o di animali da allevamento, la sfida può anche essere vinta. Ma quando l’epidemia spicca il volo, letteralmente, la circolazione del virus diventa ufficialmente fuori controllo. È quello che è accaduto con l’influenza aviaria causata da un sottotipo del virus H5N1 che dal 2020 sta facendo strage di volatili in tutto il mondo.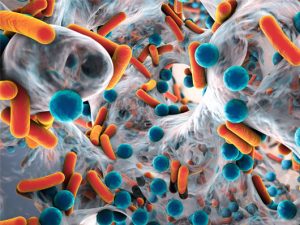 ’agenzia europea per i medicinali (Ema) ha pubblicato le
’agenzia europea per i medicinali (Ema) ha pubblicato le 